アミノ酸残基置換機能
【アミノ酸側鎖の置換】
生体内に存在するタンパク質は何らかの影響でいくつかのアミノ酸残基が変異することがあります。数残基しか変異していないタンパク質でも、その機能や活性が劇的に変化する場合があります。そのような現象を計算機で調べるためには、あらかじめ用意されているアミノ酸配列が記述されているPDBフォーマットを使用し、それに対してアミノ酸の置換を行なうのがよいでしょう。
また、PDBフォーマットを使用する場合、多くの構造はX線結晶解析実験で得られており、運動の大きいアミノ酸側鎖の一部の構造が決定されていない例があるため、不足分子を補う必要があります。
CONFLEXでは、PDBファイルに対してこれらのアミノ酸側鎖の置換や補足を行ない、その後の計算を行なうことができます。また、アミノ酸側鎖の不足情報のほかにも、主鎖の不足情報を得ることができます。
【側鎖伸張アルゴリズム】
アミノ酸側鎖の置換における不足分を補う処理と不足分子を補う処理は、同じアルゴリズムで行なっています。長い側鎖から短い側鎖に置換する場合は、長い部分を消去するだけの処理を行なっています。側鎖を伸ばすアルゴリズムでは、他の原子との衝突を回避するために、非結合原子間距離を元にねじれ角を最適化する手法をとっています。
【側鎖伸張の実行】
ここでは、不足原子を含むPDBファイルを用いて、
を紹介します。
まず、PDBサイトから、“1OHR”を検索し、そのPDBファイル(1ohr.pdb)をダウンロードします。
この構造はX線結晶解析で得られた阻害剤を含むHIV-1プロテアーゼで、99アミノ酸残基の2量体タンパク質です。この決定された構造には、「REMARK 470」で始まる行に不足原子の情報が記されています。
1OHR.pdbのREMARK 470で始まる行(不足原子の情報が記載されている)
REMARK 470 REMARK 470 MISSING ATOM REMARK 470 THE FOLLOWING RESIDUES HAVE MISSING ATOMS (M=MODEL NUMBER; REMARK 470 RES=RESIDUE NAME; C=CHAIN IDENTIFIER; SSEQ=SEQUENCE NUMBER; REMARK 470 I=INSERTION CODE): REMARK 470 M RES CSSEQI ATOMS REMARK 470 GLN A 7 CD OE1 NE2 REMARK 470 LYS A 14 CE NZ REMARK 470 GLU A 34 CD OE1 OE2 REMARK 470 GLU A 35 CD OE1 OE2 REMARK 470 ARG A 41 CG CD NE CZ NH1 NH2 REMARK 470 LYS A 43 CG CD CE NZ REMARK 470 LYS A 45 CG CD CE NZ REMARK 470 LYS A 55 CD CE NZ REMARK 470 GLN A 61 CG CD OE1 NE2 REMARK 470 LYS A 70 CE NZ REMARK 470 GLN B 7 CD OE1 NE2 REMARK 470 LYS B 14 CG CD CE NZ REMARK 470 ARG B 41 CG CD NE CZ NH1 NH2 REMARK 470 LYS B 55 CE NZ REMARK 470 GLN B 61 CD OE1 NE2
ここから、「REMARK 470 GLN A 7 CD OE1 NE2」と書かれている行は、「A鎖の7番目の残基であるグルタミンのCδ、Oε1、Nε2原子が不足している」のように読み取ることができます。ただし、CONFLEX内部では282行目以降の「ATOM」で始まる行を参照していますので、この部分を変更しても計算には影響しません。
まず、原子が不足している状態でCONFLEXを実行してみます。
[Interfaceから実行する場合]
1ohr.pdbファイルをCONFLEX Interfaceを用いて開きます。
Calculationメニューから「CONFLEX」を選択し、開いた計算設定ダイアログのをクリックし詳細設定ダイアログを開きます。
次に、詳細設定ダイアログのをクリックします。
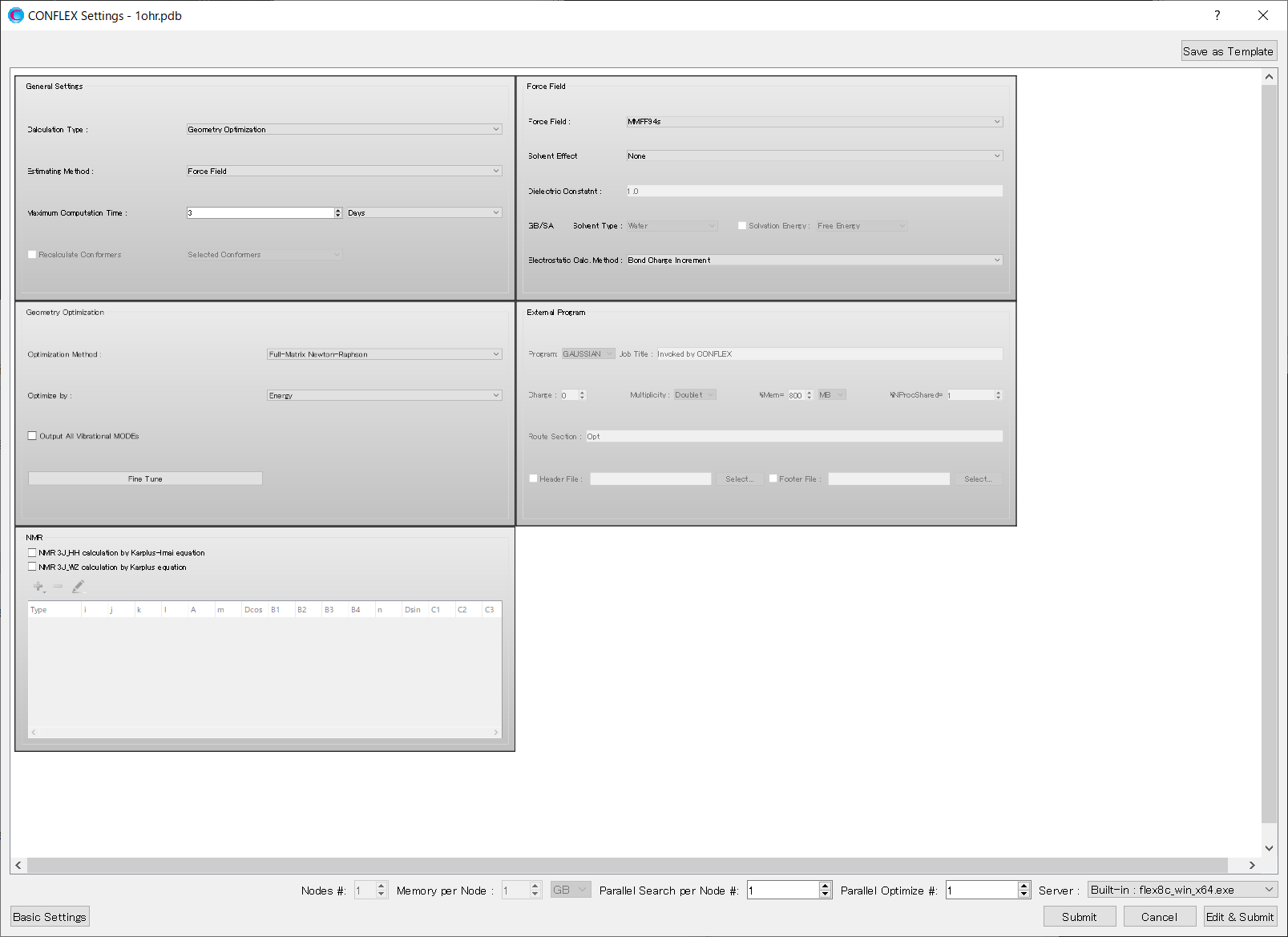
クリック後、計算設定のキーワードが記述されたダイアログが開きます。
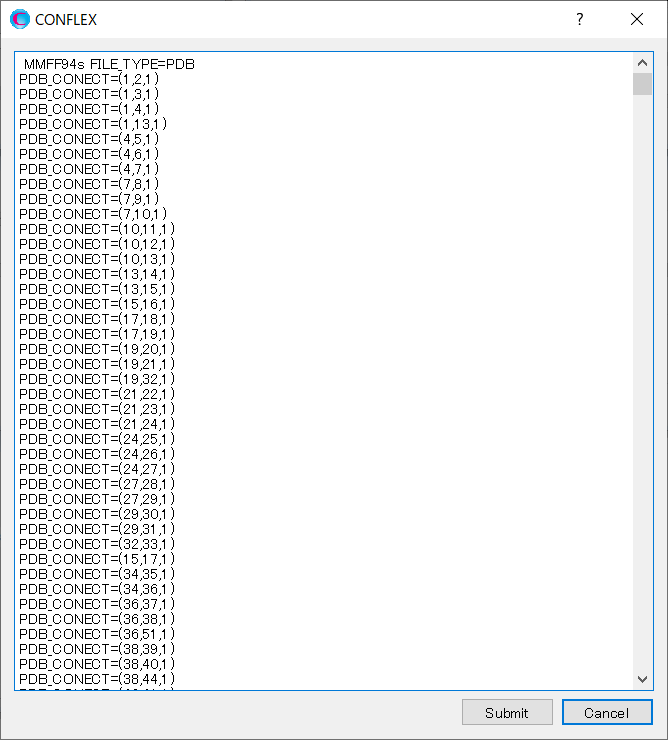
ここで、CONFLEX Interfaceが自動に「PDB_CONECT=」キーワードを使い結合情報を設定していますが、これらはすべて削除し、以下の通り阻害剤分子の二重結合の情報のみに編集します。「PDB_CONECT=(i,j,n)」において、iとjは、PDBファイルに記述されているシリアル番号、nは結合次数を表しています。
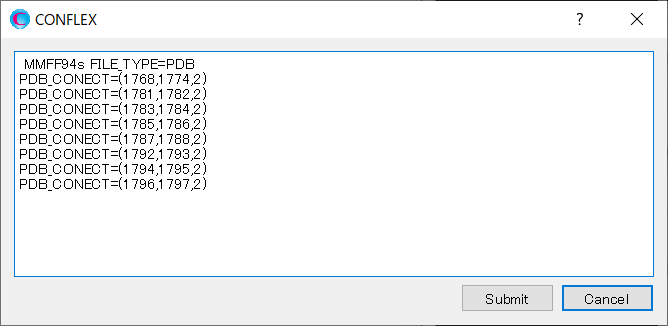 編集が終わりましたら、をクリックしてください。計算が始まります。
編集が終わりましたら、をクリックしてください。計算が始まります。
[コマンドラインから実行する場合]
計算設定は、1ohr.iniファイルにキーワードを記述することで行います。
1ohr.iniファイル
MMFF94S PDB_CONECT=(1768,1774,2) PDB_CONECT=(1781,1782,2) PDB_CONECT=(1783,1784,2) PDB_CONECT=(1785,1786,2) PDB_CONECT=(1787,1788,2) PDB_CONECT=(1792,1793,2) PDB_CONECT=(1794,1795,2) PDB_CONECT=(1796,1797,2)
「MMFF94S」は、計算にMMFF94s力場を使うことを意味します。
また、「PDB_CONECT=」は、阻害剤分子の二重結合を設定しています。「PDB_CONECT=(i,j,n)」において、iとjは、PDBファイルに記述されているシリアル番号、nは結合次数を表しています。
1ohr.pdbと1ohr.iniの二つのファイルを一つのフォルダに格納し、下記コマンドを実行してください。計算が始まります。
C:\CONFLEX\bin\conflex-10a.exe -par C:\CONFLEX\par 1ohrenter
上記は、Windowsの場合です。他のOSにおける実行コマンドについては、本書の「実行方法」を参照してください。
計算結果
CONFLEXの実行後、出力されたbsoファイルを確認すると、下記のような結果が得られ、エラー終了していることが分かります。これらは、上述した不足原子と対応していることを確認してください。
bsoファイル(原子が不足しているPDBファイルをそのまま実行した出力結果)
PDB_EXT/CHECK_AND_BUILD: ERROR -- INCLUDE UNKNOWN RESIDUE IN CHAIN A 7 *PLEASE SET KEYWORD(S) :PDB_MUTATE=(GLN,A,7) PDB_EXT/CHECK_AND_BUILD: ERROR -- INCLUDE UNKNOWN RESIDUE IN CHAIN A 14 *PLEASE SET KEYWORD(S) :PDB_MUTATE=(LYS,A,14) PDB_EXT/CHECK_AND_BUILD: ERROR -- INCLUDE UNKNOWN RESIDUE IN CHAIN A 34 *PLEASE SET KEYWORD(S) :PDB_MUTATE=(GLU,A,34) PDB_EXT/CHECK_AND_BUILD: ERROR -- INCLUDE UNKNOWN RESIDUE IN CHAIN A 35 *PLEASE SET KEYWORD(S) :PDB_MUTATE=(GLU,A,35) PDB_EXT/CHECK_AND_BUILD: ERROR -- INCLUDE UNKNOWN RESIDUE IN CHAIN A 41 *PLEASE SET KEYWORD(S) :PDB_MUTATE=(ARG,A,41) PDB_EXT/CHECK_AND_BUILD: ERROR -- INCLUDE UNKNOWN RESIDUE IN CHAIN A 43 *PLEASE SET KEYWORD(S) :PDB_MUTATE=(LYS,A,43) PDB_EXT/CHECK_AND_BUILD: ERROR -- INCLUDE UNKNOWN RESIDUE IN CHAIN A 45 *PLEASE SET KEYWORD(S) :PDB_MUTATE=(LYS,A,45) PDB_EXT/CHECK_AND_BUILD: ERROR -- INCLUDE UNKNOWN RESIDUE IN CHAIN A 55 *PLEASE SET KEYWORD(S) :PDB_MUTATE=(LYS,A,55) PDB_EXT/CHECK_AND_BUILD: ERROR -- INCLUDE UNKNOWN RESIDUE IN CHAIN A 61 *PLEASE SET KEYWORD(S) :PDB_MUTATE=(GLN,A,61) PDB_EXT/CHECK_AND_BUILD: ERROR -- INCLUDE UNKNOWN RESIDUE IN CHAIN A 70 *PLEASE SET KEYWORD(S) :PDB_MUTATE=(LYS,A,70) PDB_EXT/CHECK_AND_BUILD: ERROR -- INCLUDE UNKNOWN RESIDUE IN CHAIN B 7 *PLEASE SET KEYWORD(S) :PDB_MUTATE=(GLN,B,7) PDB_EXT/CHECK_AND_BUILD: ERROR -- INCLUDE UNKNOWN RESIDUE IN CHAIN B 14 *PLEASE SET KEYWORD(S) :PDB_MUTATE=(LYS,B,14) PDB_EXT/CHECK_AND_BUILD: ERROR -- INCLUDE UNKNOWN RESIDUE IN CHAIN B 41 *PLEASE SET KEYWORD(S) :PDB_MUTATE=(ARG,B,41) PDB_EXT/CHECK_AND_BUILD: ERROR -- INCLUDE UNKNOWN RESIDUE IN CHAIN B 55 *PLEASE SET KEYWORD(S) :PDB_MUTATE=(LYS,B,55) PDB_EXT/CHECK_AND_BUILD: ERROR -- INCLUDE UNKNOWN RESIDUE IN CHAIN B 61 *PLEASE SET KEYWORD(S) :PDB_MUTATE=(GLN,B,61)
エラーを回避し、計算を実行するためには下記の二つの方法があります。
元の座標を用いて計算を実行
一つは、そのままの座標を用いて計算を行なう方法があります。
この場合、計算設定に、「PDB_NOMUTATE」キーワードを加えます。「PDB_NOMUTATE」はアミノ酸残基中の不足している原子を加えずそのままにすることを意味します。
[Interfaceから実行する場合]
のクリック後に表示されるダイアログの内容に、「PDB_NOMUTATE」キーワードを追加します。
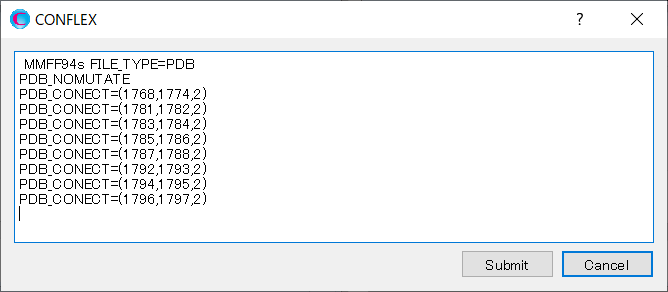 編集が終わりましたら、をクリックしてください。計算が始まります。
編集が終わりましたら、をクリックしてください。計算が始まります。
[コマンドラインから実行する場合]
1ohr.iniファイルに「PDB_NOMUTATE」キーワードを追加します。
1ohr.iniファイル
MMFF94S
PDB_NOMUTATE
PDB_CONECT=(1768,1774,2)
PDB_CONECT=(1781,1782,2)
PDB_CONECT=(1783,1784,2)
PDB_CONECT=(1785,1786,2)
PDB_CONECT=(1787,1788,2)
PDB_CONECT=(1792,1793,2)
PDB_CONECT=(1794,1795,2)
PDB_CONECT=(1796,1797,2)
1ohr.molと1ohr.iniの二つのファイルを一つのフォルダに格納し、下記コマンドを実行してください。計算が始まります。
C:\CONFLEX\bin\conflex-10a.exe -par C:\CONFLEX\par 1ohrenter
上記は、Windowsの場合です。他のOSにおける実行コマンドについては、本書の「実行方法」を参照してください。
計算結果
CONFLEXの実行後、出力されたbsoファイルを確認すると、下記のような結果が得られ、エラーメッセージが警告メッセージに変わっていることが確認できます。*大きな分子のため、計算終了には非常に時間がかかりますので注意してください。
bsoファイル(不足原子を含んだPDBファイルをそのまま実行した出力結果)
PDB_EXT/CHECK_AND_BUILD: WARNING -- INCLUDE UNKNOWN RESIDUE IN CHAIN A 7 *PLEASE SET KEYWORD(S) :PDB_MUTATE=(GLN,A,7) PDB_EXT/CHECK_AND_BUILD: WARNING -- INCLUDE UNKNOWN RESIDUE IN CHAIN A 14 *PLEASE SET KEYWORD(S) :PDB_MUTATE=(LYS,A,14) PDB_EXT/CHECK_AND_BUILD: WARNING -- INCLUDE UNKNOWN RESIDUE IN CHAIN A 34 *PLEASE SET KEYWORD(S) :PDB_MUTATE=(GLU,A,34) PDB_EXT/CHECK_AND_BUILD: WARNING -- INCLUDE UNKNOWN RESIDUE IN CHAIN A 35 *PLEASE SET KEYWORD(S) :PDB_MUTATE=(GLU,A,35) PDB_EXT/CHECK_AND_BUILD: WARNING -- INCLUDE UNKNOWN RESIDUE IN CHAIN A 41 *PLEASE SET KEYWORD(S) :PDB_MUTATE=(ARG,A,41) PDB_EXT/CHECK_AND_BUILD: WARNING -- INCLUDE UNKNOWN RESIDUE IN CHAIN A 43 *PLEASE SET KEYWORD(S) :PDB_MUTATE=(LYS,A,43) PDB_EXT/CHECK_AND_BUILD: WARNING -- INCLUDE UNKNOWN RESIDUE IN CHAIN A 45 *PLEASE SET KEYWORD(S) :PDB_MUTATE=(LYS,A,45) PDB_EXT/CHECK_AND_BUILD: WARNING -- INCLUDE UNKNOWN RESIDUE IN CHAIN A 55 *PLEASE SET KEYWORD(S) :PDB_MUTATE=(LYS,A,55) PDB_EXT/CHECK_AND_BUILD: WARNING -- INCLUDE UNKNOWN RESIDUE IN CHAIN A 61 *PLEASE SET KEYWORD(S) :PDB_MUTATE=(GLN,A,61) PDB_EXT/CHECK_AND_BUILD: WARNING -- INCLUDE UNKNOWN RESIDUE IN CHAIN A 70 *PLEASE SET KEYWORD(S) :PDB_MUTATE=(LYS,A,70) PDB_EXT/CHECK_AND_BUILD: WARNING -- INCLUDE UNKNOWN RESIDUE IN CHAIN B 7 *PLEASE SET KEYWORD(S) :PDB_MUTATE=(GLN,B,7) PDB_EXT/CHECK_AND_BUILD: WARNING -- INCLUDE UNKNOWN RESIDUE IN CHAIN B 14 *PLEASE SET KEYWORD(S) :PDB_MUTATE=(LYS,B,14) PDB_EXT/CHECK_AND_BUILD: WARNING -- INCLUDE UNKNOWN RESIDUE IN CHAIN B 41 *PLEASE SET KEYWORD(S) :PDB_MUTATE=(ARG,B,41) PDB_EXT/CHECK_AND_BUILD: WARNING -- INCLUDE UNKNOWN RESIDUE IN CHAIN B 55 *PLEASE SET KEYWORD(S) :PDB_MUTATE=(LYS,B,55) PDB_EXT/CHECK_AND_BUILD: WARNING -- INCLUDE UNKNOWN RESIDUE IN CHAIN B 61 *PLEASE SET KEYWORD(S) :PDB_MUTATE=(GLN,B,61)
側鎖を伸張して計算を実行
もう一つは、不足原子を補って計算を行う方法です。
上記した出力ファイルに「PDB_MUTATE=(GLN,A,7)」等を設定ファイルに記述するメッセージが確認できます。「PDB_NOMUTATE」を消し、これらをコピー&ペーストにより、計算設定に加えます。
「PDB_MUTATE=」キーワードは、側鎖の置換および補足を行なうキーワードで、1番目の要素は置換するアミノ酸名(1文字表記、3文字表記、フルネーム表記に対応:下表)、2番目の要素はタンパク質鎖のID、3番目の要素は残基番号を示しています。
| アミノ酸名 | 1文字 | 3文字 | フルネーム |
|---|---|---|---|
| アラニン | A | ALA | ALANINE |
| システイン | C | CYS | CYSTEINE |
| アスパラギン酸 | D | ASP | ASPARTIC_ACID |
| グルタミン酸 | E | GLU | GLUTAMIC_ACID |
| フェニルアラニン | F | PHE | PHENYLALANINE |
| グリシン | G | GLY | GLYCINE |
| ヒスチジン | H | HIS | HISTIDINE |
| イソロイシン | I | ILE | ISOLEUCINE |
| リシン | K | LYS | LYSINE |
| ロイシン | L | LEU | LEUCINE |
| メチオニン | M | MET | METHIONINE |
| アスパラギン | N | ASN | ASPARAGINE |
| プロリン | P | PRO | PROLINE |
| グルタミン | Q | GLN | GLUTAMINE |
| アルギニン | R | ARG | ARGININE |
| セリン | S | SER | SERINE |
| トレオニン | T | THR | THREONINE |
| バリン | V | VAL | VALINE |
| トリプトファン | W | TRP | TRYPTOPHAN |
| チロシン | Y | TYR | TYROSINE |
[Interfaceから実行する場合]
のクリック後に表示される起動ダイアログの内容に、「PDB_MUTATE」キーワードを追加します。
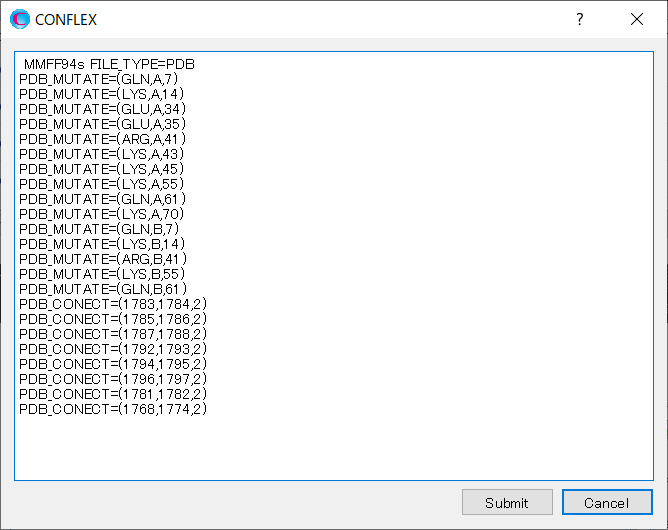 編集が終わりましたら、をクリックしてください。計算が始まります。
編集が終わりましたら、をクリックしてください。計算が始まります。
[コマンドラインから実行する場合]
1ohr.iniファイルに「PDB_MUTATE」キーワードを追加します。
1ohr.iniファイル
MMFF94S PDB_MUTATE=(GLN,A,7) PDB_MUTATE=(LYS,A,14) PDB_MUTATE=(GLU,A,34) PDB_MUTATE=(GLU,A,35) PDB_MUTATE=(ARG,A,41) PDB_MUTATE=(LYS,A,43) PDB_MUTATE=(LYS,A,45) PDB_MUTATE=(LYS,A,55) PDB_MUTATE=(GLN,A,61) PDB_MUTATE=(LYS,A,70) PDB_MUTATE=(GLN,B,7) PDB_MUTATE=(LYS,B,14) PDB_MUTATE=(ARG,B,41) PDB_MUTATE=(LYS,B,55) PDB_MUTATE=(GLN,B,61) PDB_CONECT=(1768,1774,2) PDB_CONECT=(1781,1782,2) PDB_CONECT=(1783,1784,2) PDB_CONECT=(1785,1786,2) PDB_CONECT=(1787,1788,2) PDB_CONECT=(1792,1793,2) PDB_CONECT=(1794,1795,2) PDB_CONECT=(1796,1797,2)
1ohr.molと1ohr.iniの二つのファイルを一つのフォルダに格納し、下記コマンドを実行してください。計算が始まります。
C:\CONFLEX\bin\conflex-10a.exe -par C:\CONFLEX\par 1ohrenter
上記は、Windowsの場合です。他のOSにおける実行コマンドについては、本書の「実行方法」を参照してください。
計算結果
CONFLEXの実行後、出力されたbsoファイルを確認すると、下記のような結果が得られ、置換した箇所がわかります。
bsoファイル(側鎖を補足した出力ファイル)
PDB_EXT: MUTATE RESIDUE FROM 7GLN TO GLN PDB_EXT: MUTATE RESIDUE FROM 14LYS TO LYS PDB_EXT: MUTATE RESIDUE FROM 34GLU TO GLU PDB_EXT: MUTATE RESIDUE FROM 35GLU TO GLU PDB_EXT: MUTATE RESIDUE FROM 41ARG TO ARG PDB_EXT: MUTATE RESIDUE FROM 43LYS TO LYS PDB_EXT: MUTATE RESIDUE FROM 45LYS TO LYS PDB_EXT: MUTATE RESIDUE FROM 55LYS TO LYS PDB_EXT: MUTATE RESIDUE FROM 61GLN TO GLN PDB_EXT: MUTATE RESIDUE FROM 70LYS TO LYS PDB_EXT: MUTATE RESIDUE FROM 7GLN TO GLN PDB_EXT: MUTATE RESIDUE FROM 14LYS TO LYS PDB_EXT: MUTATE RESIDUE FROM 41ARG TO ARG PDB_EXT: MUTATE RESIDUE FROM 55LYS TO LYS PDB_EXT: MUTATE RESIDUE FROM 61GLN TO GLN
不足した原子を補足する場合、PDBファイルから元の側鎖の残基名を取得しているため、元の側鎖の残基名と置換後の側鎖の残基名が同じになってしまいますが、不足原子を補足することができます。