- 製品&サービス
- CONFLEX DOCK
CONFLEX DOCKは、基質となるタンパク質に対して、指定したペプチド鎖がどこに配位し複合体を形成するかを予測するドッキングシミュレーションプログラムです。
細胞内に存在するタンパク質にとってペプチドは、インスリン等のように治療薬として直接機能する分⁄子が存在するだけでなく、 多くのタンパク質間相互作⁄用を媒介する役割を果たしていることが知られています。 またペプチドは、 合成の容易さや細胞内への浸透性等の利点があることから、中分⁄子創薬の分野でも注⁄目されている素材の⁄一つです。 タンパク質―ペプチド間の相互作⁄用の詳細を解析することで、標的タンパク質に対して有効な薬剤分⁄子を設計するための有⁄用な情報が得られることが期待できます。
p>
CONFLEX DOCKで予測する際には、タンパク質のアミノ酸残基を代表点(Cα原子)で粗視化し、ドロネー分割により四面体を構築します。
ドッキングポーズは、溶媒効果を求める際に⁄用いられる表⁄面積計算⁄手法を適⁄用して配置した、タンパク質表⁄面上の探索点を元に求めます。
そして、タンパク質表面に設定した探索点にペプチドの残基を置きます。それらの親和性の評価のため、アミノ酸残基を粗視化して実験構造データベースを元に求めた4体ポテンシャルをスコアとして⁄用いています。
従来のプログラムでは、原⁄子間相互作⁄用を元にスコアを算出することがほとんどですが、粗視化したポテンシャルを⁄用いることでスコアの算出が容易になり、巨⁄大なタンパク質でも⁄高速かつ網羅的に探索することが可能です。
他のプログラムでは、⁄入⁄力データとしてペプチドの三次元構造が必要な場合がありますが、CONFLEX DOCKの計算に必要な情報はタンパク質の三次元構造とペプチドの配列のみです。
ただし、ペプチドの構造が既知である場合、その座標データを⁄入⁄力することで探索点との差を算出し⁄比較することも可能にしています。
詳細はアルゴリズムのページをご参照ください。
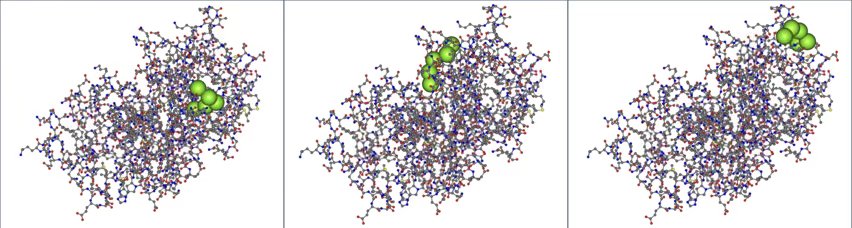
計算例として、Krev interaction trapped protein 1 (KRIT1, 4hdq.pdbのA鎖)に対する5残基のペプチド(Arg-Arg-Asp-Tyr-Phe、緑⁄色の球)のドッキングポーズの⁄一部を以下に⁄示します。スコアは、左からそれぞれ 45.96, 41.37, 38.32 です。
【文献引用】
T. Yamamoto, Y. Ikabata, H. Goto,
“Reconstruction of Four-Body Statistical Pseudopotential for Protein-Peptide Docking”,
J. Comput. Chem., Jpn.-Int. Ed., 2024, 10, 2023-0039.